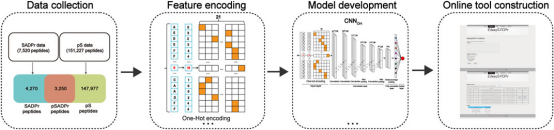
Post-translational modification (PTM) crosstalk是指发生在蛋白质同一残基上同时发生不同类型的PTM。crosstalk位点与发生一种PTM的位点具有不同的特征。针对单一PTM的研究众多,而针对crosstalk位点的研究却寥寥无几。近日,基础医学院生物信息团队于《Frontiers in Cell and Developmental Biology》(中科院2区, IF=6.081)发表了题为“EdeepSADPr: an extensive deep-learning architecture for prediction of the in situ crosstalks of serine phosphorylation and ADP-ribosylation”的研究成果。基础医学院生物信息学研究生姜浩强、特种医学系商士朋助理教授为第一作者,李磊教授为通讯作者。该研究受到国家自然科学基金(32071430,32271504)和山东省科技型中小企业创新能力提升工程(2021TSGC1295)资助。
在这项研究中,构建了能够分别从serine phosphorylation数据集、serine ADP-ribosylation数据集和蛋白质序列中预测同时发生phosphorylation和ADP-ribosylation修饰的serine位点(pSADPr)分类器,并使用十倍交叉验证和独立测试数据集中评估了五个深度学习分类器。我们还使用以上分类器作为基本分类器来开发集成分类器以提高性能。从ADP-ribosylation、phosphorylation和未修饰的丝氨酸位点中识别pSADPr位点的最佳分类器AUC分别为0.700、0.914和0.954。最后,团队基于CNNOH分类器开发了能够预测人类pSADPr位点的在线工具(EdeepSADPr)。它可以通过http://edeepsadpr.bioinfogo.org/免费获得。